Náš neustálý zájem nalézt nejvhodnější technologická řešení pro Vaše experimenty nás dovedl k zavedení technologie GemCode do naší nabídky služeb v oblasti Next-Generation sekvenování. Tímto příspěvkem hrdě oznamujeme dostupnost této technologie v naší laboratoři a zároveň představujeme její základní vlastnosti.
GemCode technologie v podobě, jak ji zde popisujeme, byla rozvinuta společností 10xGenomics se sídlem ve městě Pleasanton (již dle názvu příjemné místo) v Kalifornii. Tato technologie dala vzniknout systému přístrojů, reagencií a analytických programů nazývanému - Chromium system.
Hlavní přednosti systému Chromium:
Na obrázku níže jsou uvedeny příklady některých aplikací (Zheng et al. 2017):
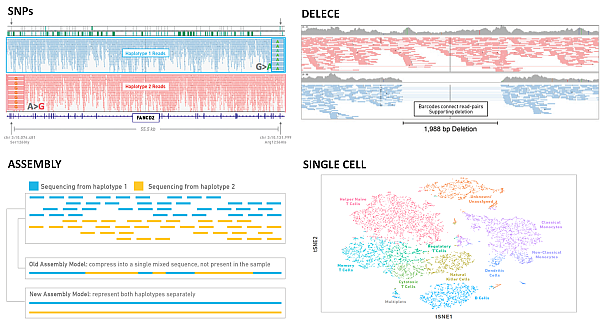
Technologie
Technologie GEM („Gel bead in Emulsion“) umožňuje uzavřít dlouhé molekuly DNA nebo RNA z jediné buňky do emulzní kapky společně s gelovou partikulí („gel bead“) nesoucí barkódované primery. Pomocí těchto primerů se v partikuli připraví krátké fragmenty s barkódem k sekvenaci. Primery nesoucí barkódy jsou specifické pro každou jednotlivou partikuli, což umožní zpětně zrekonstruovat zdrojovou molekulu DNA (DNA aplikace) nebo identifikovat transkripty pocházející z jedné buňky (RNA aplikace).
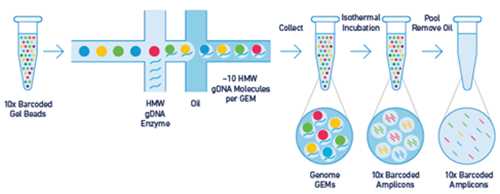
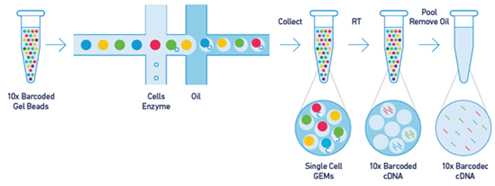
Řešení pro genomické aplikace (vlevo) a řešení pro transkriptomiku (vpravo)
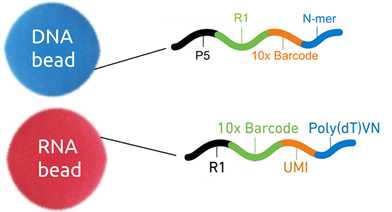
Jedním z klíčových prvků GemCode technologie jsou primery asociované s partikulemi. Tyto primery jsou složeny z části zajišťující nasednutí na cílovou molekulu – N-mer pro DNA aplikace a polyT úsek pro RNA aplikace – 14-bázový barkód a sekvenci pro nasednutí sekvenačního primeru (kompatibilní pouze s přístroji Illumina). Primery pro DNA navíc nesou sekvenci pro hybridizaci na čip a primery pro RNA nesou navíc sekvenci UMI („Unique Molecular Identifier“), kterou lze využít při analýze dat k redukci kvantitativních nepřesností zanesených při amplifikaci templátů.
Přístrojové vybavení
Příprava sekvenační knihovny s použitím technologie GEM se provádí v přístroji „Chromium controller“, který by šlo nejlépe charakterizovat jako kombinaci průtokového cytometru a emulzního PCR cykleru.

„Chromium controller“ využívá až 750 000 různých barkódů. V každé emulzní kapénce se zachytí několik molekul, což dává možnost rekonstrukce přibližně 5 – 10 miliónů 50 kb sekvencí, nebo možnost analýzy transkriptomu několika stovek tisíc jednotlivých buněk.
Technologie GemCode výrazně posouvá možnosti NGS sekvenování a otevírá přístup k informacím, ke kterým byste se jinak se sekvenátory generujícími krátké sekvence nedostali. Zapojení této technologie nevyžaduje žádné velké změny v plánování experimentu a je plně kompatibilní se sekvenátory Illumina. Od začátku pracujete se stejným materiálem jako při "běžných" NGS experimentech, ale získáte data poskytující mnohem více informací než data získaná s konvenční sekvenační knihovnou.
Analýza dat
Zásadním krokem analýzy dat sekvenačních knihoven připravených s využitím technologie GemCode je rekonstrukce zdrojových molekul. K tomuto účelu společnost 10x Genomics poskytuje volně přístupný software Long Ranger. Další programy pro následnou analýzu dat – Supernova pro de-novo sestavování genomů, Cell Ranger pro analýzu traskriptomu nebo Loupe pro vizualizaci dat jsou rovněž poskytnuty volně k instalaci a užívání. Součástí těchto programů je mimo jiné aligner 10x Lariat speciálně vyvinutý a vyladěný na data, kde si sekvence nesou informaci o společné zdrojové molekule.
Technické specifikace:
Pokud Vás možnosti této technologie zaujaly, tak nás prosím neváhejte kontaktovat.
NGS Lab, ngs@seqme.eu
Literatura:
Zheng GX, Terry JM, Belgrader P, et al. Massively parallel digital transcriptional profiling of single cells. Nat Commun. 2017;8:14049.